2022年1月17日,我校棉花遗传改良团队联合美国农业部南方研究中心在开放获取期刊Communications Biology发表了题为“Genomic interrogation of a MAGIC population highlights genetic factors controlling fiber quality traits in cotton”的研究论文。该研究基于550个重组自交系构成的MAGIC(multi-parent advanced generation intercross)群体,结合7年12个环境的表型数据全面剖析了陆地棉纤维品质性状的遗传基础。
棉花是世界上最重要的天然纺织纤维来源。充分理解棉花纤维品质性状的遗传基础,加快选育优质的棉花新品种对棉花产业的可持续发展具有非常重要的意义。传统上,研究人员主要通过全基因组关联分析(GWAS)利用自然群体解析纤维品质性状的遗传基础。但由于自然群体存在明显的群体结构、未知的亲缘关系及普遍的低频功能变异,针对复杂性状的遗传定位效果并不理想。相较于自然群体,MAGIC群体没有这些劣势,能够有效地控制关联分析的假阳性。
本研究通过对11个亲本及550个子代进行全基因组测序,共获得了1,548,294个高质量的SNP,并在此基础上利用隐马尔可夫模型进行了血缘同源(Identity By Descent,IBD)分析,总共鉴定出115,316个IBD片段(图1)。通过基于SNP和单倍型(IBD)的全基因组关联分析,研究人员鉴定出与纤维品质(纤维长度、强度、伸长率和整齐度)相关的25个sQTL和14个hQTL,其中26个为新发现的QTL。两种GWAS方法共同检测到了两个主效QTL,分别与纤维强度和长度有关。一个编码MATE家族蛋白的基因(GhZF14)被确定为控制纤维长度的新候选基因。此外,研究人员还鉴定出581个显著的上位性互作,平均每对互作可以解释4%的表型变异。
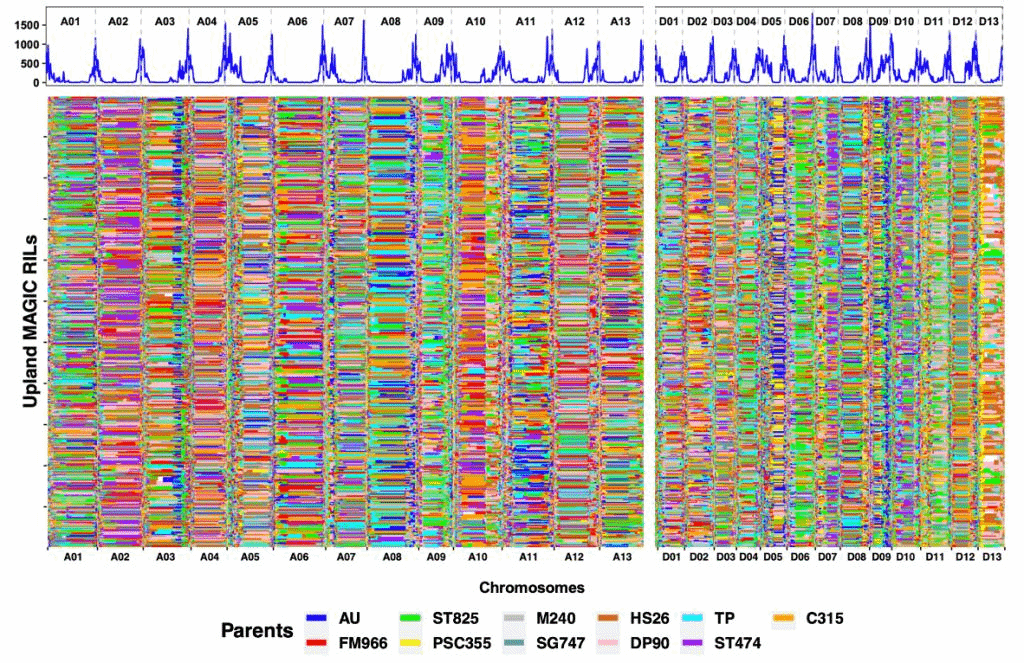
图一 550个MAGIC自交系的IBD片段分布
我院王茂军教授、博士研究生戚正阳为文章的共同第一作者,张献龙教授和美国农业部南方研究中心的David Fang研究员为本文的通讯作者。
论文链接:https://www.nature.com/articles/s42003-022-03022-7


